自然微生物组是生物修复的重要资源,能够直接参与高分子量多环芳烃的降解。稳定性同位素核酸探针技术(DNA - stable isotope probing,DNA-SIP)能够在群落水平上将微生物群落结构与功能联系起来直接揭示复杂环境中功能微生物的分子机制。我室滕应研究员课题组前期研究发现由红壤、水稻土构建的复合微生物组能够显著促进红壤中芘的去除,但未能对其中的功能微生物直接进行识别。因此本课题组进一步对红壤、水稻土微生物群落以及二者混合后构建的复合微生物组(PR)中的苯并[a]芘(BaP)活性降解微生物进行鉴别。
研究发现,土壤中BaP的去除只要依赖于微生物活动,同时PAH环羟基化双加氧酶(PAH-RHD)基因的表达量与BaP去除效果显著正相关。此外,红壤(R)中BaP的去除效率最低,标记到的活性降解功能的微生物仅有OTU830(Ramlibacter)。水稻土(P)中BaP的去除效率高达29.5%,被13C标记到的活性BaP降解菌种类最多。在复合微生物组PR中BaP的去除效率为25.3%,仅次于P;被13C标记到的功能微生物除了Nocardioides和Micromonospora与P中的相同之外,还有Burkholderia和Phenylobacterium。这表明复合微生物组中的活性降解菌与BaP去除潜力较强的水稻土群落有一致性,同时也具有其独特性。本研究基于DNA-SIP技术首次直接鉴别到具有BaP降解活性的微生物有Saccharothrix、Phenylobacterium、Micromonospora和Nocardioides(图1)。基于功能预测,对土壤中BaP降解相关的功能基因进行分析,并构建了土壤中BaP的可能代谢路径(图2)。
相关成果发表在Journal of Hazardous Materials期刊上发表,博士生王贝贝为论文第一作者,滕应研究员为论文通讯作者,该研究工作得到江苏省优秀青年基金等资助。

图1 土壤中利用13C标记BaP的降解性微生物种类
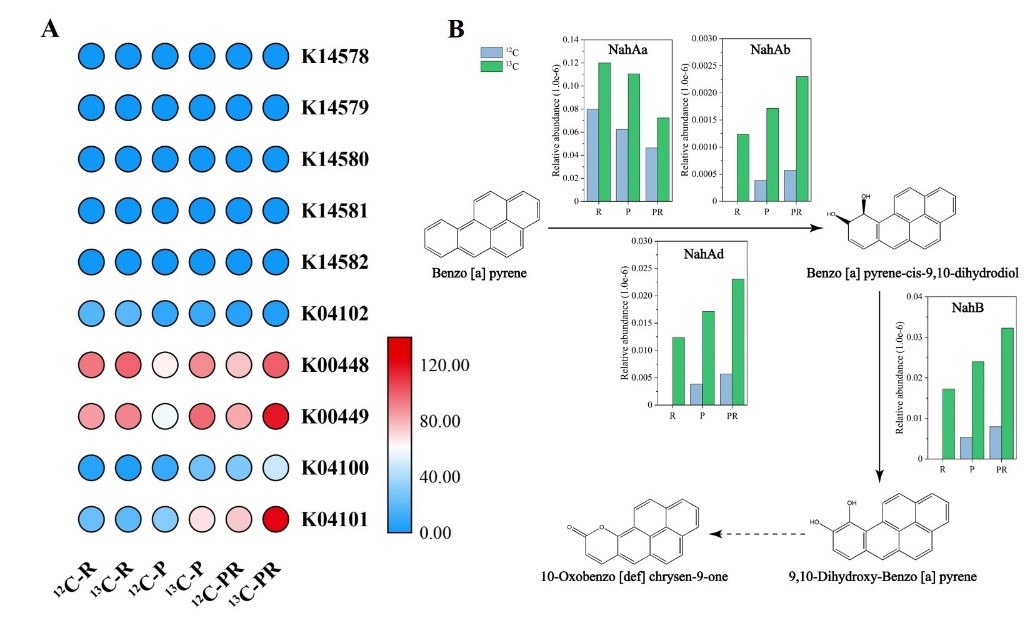
图2 Tax4Fun预测中与PAH代谢途径相关的KOs丰度热图(A)和基于功能预测构建的土壤中BaP的可能代谢路径(B)